L’AMPÉLOGRAPHIE MOLÉCULAIRE
Les techniques modernes de génotypage et d’analyse de l’ADN nucléaire et chloroplastique ont révolutionné l’ampélographie. Elles permettent d’étudier les relations phylogénétiques entre espèces, de différencier les grands groupes au sein de l’espèce Vitis vinifera et de distinguer les variétés. Elles rendent également possible la recherche de parentés entre cépages, et l’élaboration de véritables arbres généalogiques qui ont mis en lumière le rôle central de certains grands géniteurs dans les encépagements autochtones.
Qu’est-ce que l’ampélographie moléculaire ?

Le terme ampélographie vient du grec «ampelos» qui signifie vigne et de «graphie» qui se rapporte à la description. Proposé pour la première fois en 1661 par le Docteur Sachs dans l’ouvrage Ampelographia, ce terme regroupe aujourd’hui à la fois la description et l’identification des cépages, l’étude de leur évolution et des relations qui existent entre eux et la connaissance de leur comportement vis à vis du milieu (aptitudes culturales et œnologiques). Pendant longtemps, l’ampélographie ne s’est basée que sur l’observation visuelle de la vigne, en particulier sur la morphologie des feuilles, des rameaux et des grappes. Parmi une centaine de descripteurs codifiés pour décrire les différents organes de la vigne, 14 descripteurs primaires prioritaires ont été retenus par l’OIV pour leur bon pouvoir discriminant entre les variétés. Plus récemment, l’ampélographie s’est dotée de nouveaux outils comme la chimiotaxonomie (études des composés du métabolisme secondaire) ou les marqueurs biochimiques (isozymes). Ces marqueurs ont cependant montré leurs limites, leur pouvoir discriminant s’étant avéré insuffisant pour un usage à grande échelle. La véritable révolution est née de l’incorporation à l’ampélographie de techniques génétiques à partir des années 1990. Le marquage moléculaire et le séquençage de l’ADN ont permis d’avoir accès non seulement au phénotype mais aussi directement au génotype de façon précoce sans avoir besoin d’attendre un développement complet de la plante. L’ADN nécessaire pour ce type d’analyse peut être extrait à partir de quelques dizaines de milligrammes de matériel végétal et de n’importe quel organe ou partie de la plante (feuilles, baies, sarments, racines…).
Qu’est-ce que l’ADN et comment est-il structuré ?
L’acide désoxyribonucléique, ou ADN, est une molécule, présente dans toutes les cellules vivantes, qui renferme l’ensemble des informations nécessaires au développement et au fonctionnement d’un organisme. C’est aussi le support de l’hérédité car il est transmis lors de la reproduction, de manière intégrale ou non. Il porte donc l’information génétique et constitue le génome des êtres vivants. L’ADN détermine la synthèse des protéines, par l’intermédiaire de l’ARN. Cette double hélice est composée de séquences de nucléotides constitués d’un groupe phosphate lié à un sucre, le désoxyribose, lui-même lié à une base azotée. Il existe quatre bases azotées différentes : la cytosine (notée C) et la thymine (notée T) de la famille des pyrimidines, et l’adénine (notée A) et la guanine (notée G) de la famille des purines. Dans les cellules végétales, l’ADN est localisé dans le noyau, dans les chloroplastes et dans les mitochondries. Les chloroplastes sont des organites essentiels qui permettent de capter la lumière à l’origine de la photosynthèse. Les mitochondries sont impliquées dans les processus de fourniture d’énergie à la cellule.
A quelles techniques l’ampélographie moléculaire fait-elle appel ?
L’ampélographie moléculaire fait appel à l’analyse de plusieurs types de marqueurs sur l’ADN, qui permettent de différencier les individus avec certitude :
- les microsatellites (nucléaires et chloroplastiques) ou SSR (Simple Sequence Repeats) : Il s’agit de courtes séquences d’ADN formées de la répétition (jusqu’à 20 à 30 fois) d’unités de 2,3 ou 4 bases (par exemple C-A ou G-A-T).
- les indels qui sont des mutations d’insertion ou de délétion de courtes séquences d’ADN
- les éléments transposables ou éléments mobiles parfois aussi nommés «gènes sauteurs». Leurs déplacements peuvent être activés par les stress et certains facteurs environnementaux. La mobilité de ces éléments est une source de mutations, donc de variabilité génétique et d’évolution. On distingue les rétrotransposons qui se répandent selon un mode «copier-coller» et les transposons qui se déplacent par «couper-coller»
- les polymorphismes de nucléotides uniques ou SNP (Single Nucleotid Polymorphism) qui ne concernent qu’un seul nucléotide. Il s’agit d’un changement d’une base par une autre. On distingue les transitions qui sont des substitutions de nucléotides au sein d’une même famille (purine ou pyrimidine) et les transversions, changements d’une purine en une pyrimidine ou inversement.
Qu’a permis d’apprendre l’ampélographie moléculaire sur l’origine de la vigne?
Les travaux réalisés entre autres par l’équipe de Jean-Pierre Péros de l’INRA de Montpellier ont permis de mettre en évidence le caractère ancestral des espèces de vigne asiatiques qui seraient à la fois à l’origine de l’espèce européenne Vitis vinifera et des espèces américaines. En ce qui concerne ces dernières, il se serait produit deux événements distincts de dispersion depuis l’Eurasie vers l’Amérique : un événement à l’origine des espèces du centre et de l’Est (Vitis labrusca, Vitis riparia, Vitis rupestris et Vitis berlandieri), l’autre à l’origine des espèces californiennes (Vitis californica). L’analyse des micro-satellites a permis également de différencier les vraies vignes sauvages européennes (Vitis vinifera subsp. sylvestris) appelées aussi lambrusques des variétés cultivées. Au sein de ce compartiment cultivé, ces analyses ont permis de répartir objectivement les variétés en cinq grands groupes géographiques en relation avec leurs aptitudes.
Qu’a permis d’apprendre l’ampélographie moléculaire sur les liens de parenté entre cépages ?
L’analyse par micro-satellites des 2300 cépages traditionnels conservés au Domaine de Vassal (INRA de Marseillan) a permis de constituer une base de données complète et unique dans son genre. Lorsque sur l’ensemble des marqueurs d’une variété étudiée, on peut trouver par comparaison deux autres cépages ayant chacun la moitié des valeurs déterminées, il existe une forte probabilité que par croisement ces deux cépages aient donné naissance au troisième. Il est même parfois possible de déterminer pour un croisement donné quel est le père d’où provient le pollen et la mère qui a porté le fruit. En effet, c’est la mère qui fournit au futur descendant, l’ensemble de sa «machinerie métabolique» en particulier ses chloroplastes. Ces derniers contiennent également de l’ADN sur lequel on a pu déterminer certains marqueurs génétiques.
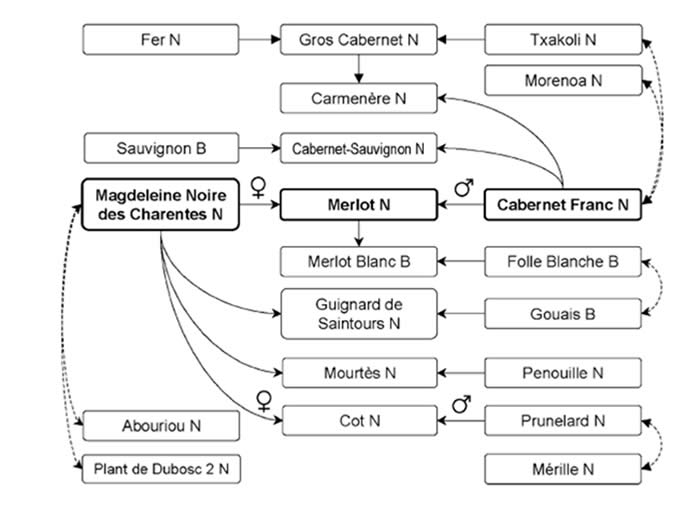
Il a ainsi été montré que le Gouais B, variété blanche sans grand intérêt œnologique, était un géniteur important de nos cépages. Par exemple, par croisement avec un Pinot, il a engendré le Chardonnay B, l’Aligoté B, l’Auxerrois B, le Melon B et le Gamay N. Avec d’autres parents souvent inconnus, il est également à l’origine entre autre de la Jacquère B, du Grolleau N, du Colombard B, du Riesling B ou même du Saint-Côme B aveyronnais. Plus récemment, une étude menée par Jean-Michel Boursiquot et d’autres collaborateurs, publiée fin 2008 dans une revue scientifique australienne, a permis d’identifier la parenté du Merlot N. Lors de prospections réalisées dans des vieilles parcelles de vignes en Bretagne et dans les Charentes, un cépage original et inconnu des collections a pu être identifié. En raison de sa précocité, cette variété était nommée « Magdeleine » par certains propriétaires qui la cultivaient en treille (la Sainte Madeleine tombe le 22 juillet). Elle fut alors dénommée « Magdeleine noire des Charentes » pour la distinguer des autres variétés (surtout de table) déjà dénommées Madeleine. Depuis, ce cépage a également été retrouvé à 3 reprises sous forme de treilles dans le département du Gers, chaque fois sous le nom de Madeleine, ce qui constitue un fait assez remarquable. Les analyses génétiques ont confirmé que cette variété, probablement très ancienne et quasiment absent des textes ampélographiques, était la mère du Merlot N et du Cot N (Malbec). On savait également que le Prunelard N et le Malbec étaient apparentés sans connaître jusque là le sens de filiation. On sait dorénavant que le Prunelard N est le père du Malbec. Le schéma ci-dessous (figure 1) issu de la publication illustre quelques liens de parenté entre les cépages.
Quels sont les exemples les plus marquants d’identification variétale réussie à l’aide de l’ampélographie moléculaire ?
En 2009, Jean-Michel Boursiquot et Laurent Audeguin du Pôle Matériel Végétal de l’IFV, ont pu mettre en évidence, un faux Albarinho B lors d’une mission en Australie. Depuis de nombreuses années, les viticulteurs locaux croyaient cultiver cette variété originaire de la façade ouest de la péninsule ibérique, mais nos collègues ont rapidement déterminé qu’il s’agissait en fait du Savagnin B. Ce résultat a été confirmé peu de temps après leur retour en France, grâce à l’analyse génétique d’ADN prélevé sur place et après confrontation aux bases de données existantes (IFV, INRA). Pour l’anecdote, la même histoire s’était déjà produite au Chili près de vingt ans auparavant et bien avant l’utilisation des marqueurs génétiques. La Carmenère N y avait longtemps été confondue par les viticulteurs avec le Merlot N. Dans la région, ces techniques ont permis à l’IFV Sud-Ouest d’identifier toutes les anciennes variétés maintenues dans les conservatoires régionaux, et d’introduire dans les collections des cépages originaux qui n’y figuraient pas tels le Moural N de l’Aveyron, le Bouysselet B de Fronton et d’autres variétés sans dénomination à ce jour.
L’ampélographie moléculaire permet-elle de distinguer les clones ?
Aujourd’hui, sauf quelques cas particuliers (voir plus bas), l’ampélographie moléculaire ne permet pas de distinguer les clones d’un même cépage, car il existe très peu de différence intra-variétale au niveau des micro-satellites. Cependant, les nouvelles méthodes de séquençage de l’ADN à très haut débit permettent maintenant d’envisager le reséquençage du génome de différents individus. Cette approche a été choisie pour essayer de détecter les différences génétiques pouvant exister entre clones. Au cours d’une thèse menée par Grégory Carrier dans le cadre de l’UMT Géno-Vigne® et encadrée par l’IFV, Montpellier SupAgro et l’INRA, trois clones de Pinot Noir ont été analysés. Les résultats ont permis de mettre en évidence le rôle prépondérant des éléments mobiles (rétro-transposons) dans le polymorphisme génétique détecté entre ces clones. Sur cette base, une analyse préliminaire de diversité à l’aide de marqueurs SSAP a permis d’obtenir des profils uniques pour tous les clones de Pinot Noir en collection. Cependant, une sélection de ces marqueurs et une validation de leur stabilité et de leur répétabilité doivent encore être réalisées avant de pouvoir envisager une identification fiable des différents clones. D’autres travaux ultérieurs ont également permis de progresser dans la voie de l’identification des clones. Aujourd’hui, on sait par exemple caractériser, grâce à un marqueur microsatellite spécifique, la sensibilité ou non des clones de Syrah au dépérissement particulier qui touche ce cépage. Très récemment, des auteurs ont proposé un nouveau set de marqueurs microsatellites susceptibles de déterminer des mutants de couleur au sein d’une même variété, ce que ne permettaient pas les marqueurs développés jusqu’alors (Migliaro & al., 2017).
CONTACT
 Olivier Yobregat
Olivier Yobregat
olivier.yobregat@vignevin.com
Expert national : Jean-Michel Boursiquot (Montpellier SupAgro), Directeur Scientifique et Technique Matériel Végétal
POUR EN SAVOIR PLUS
Articles
- Parentage of Merlot and related winegrape cultivars of southwestern France: discovery of the missing link
- Distinguishing Carmenère from Similar Cultivars by DNA Typing
- Migliaro D., De Nardi B., Vezzulli S., Crespan M.,2017 . An Upgraded Core Set of 11 SSR Markers for Grapevine Cultivar Identification: The Case of Berry Color Mutants. American Journal of Enology and Viticulture, 2017, 68(4)
- Thierry Lacombe, Jean-Michel Boursiquot, Valérie Laucou, Manuel Di Vecchi-Staraz, Jean-Pierre Péros, Patrice This. Large-scale parentage analysis in an extended set of grapevine cultivars (Vitis vinifera L.). Theoretical and Applied Genetics, Vol.126, n°2, 401-414.